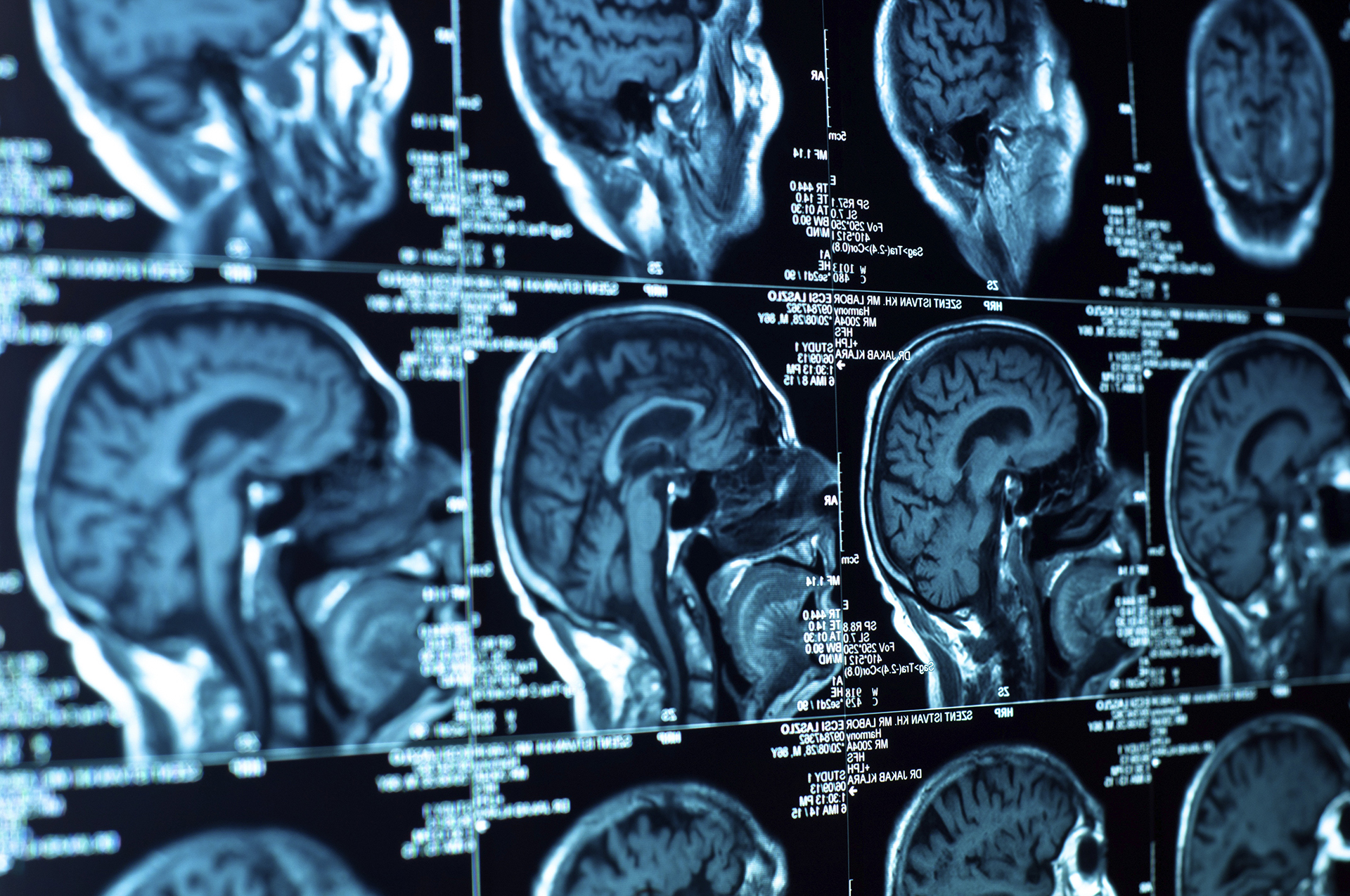
Vers le traitement personnalisé des patients atteints d’une tumeur cérébrale. Aider à améliorer le traitement du cancer du cerveau !
Cliquez sur les projets pour plus d’informations.
August 2021 – August 2024
Project PI: Michel Mittelbronn, MD, PHD
National Center of Pathology (NCP) & Luxembourg Center of Neuropathology (LCNP)
Laboratoire National de Santé
Abstract:
Radiographic imaging in glioma is routinely recorded and provides a non-invasive clinical measure before tumor resection. It is further used to monitor patients throughout their treatment and follow-up and can help to identify early relapse risk. Current standard-of-care (SOC) is neurosurgical resection, as radically as possible, radiotherapy (RT) and oral chemotherapy using temozolomide (TMZ). Guidelines for therapy focus on first-line therapy only. However, timing of second-line interventions has very important implications: Waiting too long can potentially result in tumor growth into brain areas making additional neurosurgical intervention necessary. Intervening too early, or treating pseudo-progression, exposes patients to morbidity risk and reduces quality of life.
The Mi*EDGE project is based on existing mathematical models of glioma invasion that predict tumor cell behavior based on critical density (“allee-effect”) and local metabolic-driven phenotypic plasticity (“go or grow” decision). It is aiming at predicting whether malignant cells at the margins of surgical resection transform into diffusely invasive or tumor-forming proliferative phenotypes by integrating clinical imaging and molecular pathology data, e.g. DNA methylation, multiplex-histology.
Thus, the initial investigations of the Mi*Edge consortium will be based on analysing the imaging time-points prior to operation, follow-up examinations after operation throughout the course of the disease and will include monitoring of disease progression involving MR images after re-surgery, re-RT or re-challenge with chemotherapy. We will test whether pre-surgical neuroimaging, when included into those models, can predict biological behavior of cells at the tumor edges, and if imaging after primary therapy will reflect the prediction with either diffuse tumor invasion, local relapse, or reactive changes (pseudo-progression). A number of specialized MRI scan components, such as perfusion weighted imaging (DSC-MRI), diffusion weighted or tensor imaging (DWI/ DTI) and specific contrast enhancement (DCE) will further help Mi*Edge to differentiate between unequivocal tumor progression and pseudoprogression.
Patient selection criteria and data collected:
Patients diagnosed with gliomas. The MRI scan types collected: i) Baseline MRI parameters, e.g. pre-/post-contrast T1-weighted, T2-weighted and T2-FLAIR and ii) Advanced MRI sequences, e.g. DSC-MRI, DWI/ DTI, DCE-MRI. MRI scans are anonymized by deidentification of textual data and brain extraction in the acquired cranial scans, preventing facial reconstruction/recognition.
April 2021 – ongoing
Project PIs: Simone Niclou, PHD, Anna Golebiewska, PHD & Olivier Keunen, PHD
NORLUX Neuro-Oncology laboratory and Translational Radiomics group
Department of Cancer Research
Luxembourg Institute of Health (LIH)
Abstract:
Radiographic imaging in glioma is routinely acquired, often providing the first non-invasive clinical impression (prior to biopsy or excision) and offering continuous monitoring throughout treatment and follow-up. The initial goals of iGLASS are based on the imaging timepoints, i.e., baseline pre-operative and follow-up examinations performed throughout the course of disease. At baseline, iGLASS will investigate i) distinct spatial tumor distributions of molecular characteristics, ii) mutational burden, and iii) tumor propensity to hypermutation following DNA-alkylating chemotherapy. Follow-up imaging studies linked to clinical annotation or recurrent tissue samples with molecular profiling will support investigation of i) radiogenomic biomarkers downstream of treatment, ii) longitudinal molecular alterations, and iii) treatment-induced hypermutation changes associated with worse outcome. To achieve this, iGLASS focuses on available imaging data corresponding to existing clinical and molecular data of the GLASS consortium contributors (https://glass-consortium.org/).
Patient selection criteria and data collected:
Patients diagnosed with gliomas that underwent two or more consecutive operations and were included in the GLASS consortium cohort for molecular analysis. The imaging data (MRI, CT, PET scans) will be collected and pseudonymised. The MRI scan types collected: i) Baseline MRI parameters, e.g. pre-/post-contrast T1-weighted, T2-weighted and T2-FLAIR and ii) Advanced MRI sequences, e.g. DSC-MRI, DWI/ DTI, DCE-MRI. MRI scans are anonymized by deidentification of textual data and brain extraction in the acquired cranial scans, preventing facial reconstruction/recognition.
Project website: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7566469/
April 2021 – December 2025
Project PIs: Olivier Keunen, PHD & Simone Niclou, PHD
NORLUX Neuro-Oncology laboratory and Translational Radiomics group
Department of Cancer Research
Luxembourg Institute of Health (LIH)
Abstract:
Radiographic imaging in glioma is routinely acquired throughout the management of this deadly disease. Proper detection and delineation of the tumor are important to establish diagnostic, assess the extent of the disease, plan therapy, notably radiotherapy, and assess the effect and efficacy of treatments.
The Federated Tumor Segmentation project addresses the challenging task of automatically delineating glioma, at the time of diagnosis and throughout the treatment. The task is rendered difficult by the heterogeneity of tumors that come in a variety of shapes and compositions. Artificial intelligence can help in this regard, but for the models to learn enough to reliably delineate tumor sub-regions, they need to see a large number of cases. Collecting large cohorts of patient imaging data is however difficult because of associated confidentiality and legal restrictions. So, to address this challenge, the project uses an innovative concept called Federated Learning, through which efficient models can be trained without the need to share data. With this approach, a basic model is first trained centrally before it is distributed to all participating hospitals sites. The remote sites assess the performance of the model on their local data and send the aggregator the numerical data needed to improve the model iteratively, but without ever sharing the original imaging data.
Patient selection criteria and data collected:
Patients diagnosed with gliomas for which pre-operative and follow-up MRI scans have been collected. The MRI scan types collected are baseline and follow-up scans, including pre-/post-contrast T1-weighted, T2-weighted and T2-FLAIR. Scans are anonymized by de-identification of textual data and brain extraction in the acquired cranial scans, preventing facial reconstruction/recognition.
Project website: https://www.nature.com/articles/s41467-022-33407-5
Feb 2022 – Dec 2024
Project PI: Alessandro Michelucci, PHD
Neuro-Immunology Group
Department of Cancer Research
Luxembourg Institute of Health (LIH)
Abstract:
Glioblastoma is an incurable malignant brain tumour that urgently needs new diagnostic and therapeutic approaches. A key challenge for clinical management of Glioblastoma is its highly heterogeneous nature, with heterogeneity between patients and within a single tumour representing important barriers for current therapies. The interactions between neoplastic cells and their microenvironment, including immune cells, affect Glioblastoma growth, patient survival, and response to therapy. Among non-neoplastic cells, immune cells called tumour-associated microglia/macrophages, which are mainly either resident parenchymal microglia or peripheral-monocyte derived cells, represent the majority of cells in the tumour microenvironment. Monocytes recruited from the bloodstream to the tumour site are associated with a worse prognosis compared to brain tumours with low peripheral immune cell infiltration. As tumours progress to malignancy, mediators produced by neoplastic cells and the tumour microenvironment are released into the bloodstream. These factors induce the mobilization, education and recruitment of peripheral monocytes to the tumour site, which differentiate into macrophages and stimulate angiogenesis, enhance tumour cell migration and suppress anti-tumour immunity. Further, these mediators induce severe systemic immunosuppression where, for example, peripheral lymphocytes display prominent dysfunctions and myelopoiesis is redirected from its normal pathway. Considering the tight crosstalk between the tumour in the brain and the immune components in the circulation, we here aim to analyse the blood immune cellular constituents of Glioblastoma patients, with a special focus on the monocytic populations. Taken together, the immunological profile of the blood in patients with Glioblastoma represents a signature of the tumour, which may serve for diagnosis, patient classification, prediction of immunotherapy efficacy and ultimately the discovery of novel targets.
Patient selection criteria and data collected:
Patients diagnosed with glioblastomas. The use of immune cells isolated from patients’ blood for molecular analysis.
Project website: https://www.lih.lu/en/research-scope/research-department/department-of-cancer-research/neuro-immunology-group/
Veuillez noter que le contenu n’est disponible qu’en anglais.
| Cookie | Durée | Description |
|---|---|---|
| _GRECAPTCHA | 5 mois 27 jours | Ce cookie est défini par le service recaptcha de Google pour identifier les bots afin de protéger le site Web contre les attaques malveillantes de spam. |
| cookielawinfo-checkbox-advertisement | 1an | Défini par le plugin GDPR Cookie Consent, ce cookie est utilisé pour enregistrer le consentement de l'utilisateur pour les cookies de la catégorie " Publicité " . |
| cookielawinfo-checkbox-analytics | 1an | Défini par le plugin GDPR Cookie Consent, ce cookie est utilisé pour enregistrer le consentement de l'utilisateur pour les cookies de la catégorie "Analytics" . |
| cookielawinfo-checkbox-functional | 1an | Le cookie est défini par le plugin de consentement aux cookies GDPR pour enregistrer le consentement de l'utilisateur pour les cookies de la catégorie "Fonctionnel". |
| cookielawinfo-checkbox-necessary | 1an | Défini par le plugin GDPR Cookie Consent, ce cookie est utilisé pour enregistrer le consentement de l'utilisateur pour les cookies de la catégorie " Nécessaire " . |
| cookielawinfo-checkbox-others | 1an | Défini par le plugin GDPR Cookie Consent, ce cookie est utilisé pour stocker le consentement de l'utilisateur pour les cookies de la catégorie " Autres ". |
| cookielawinfo-checkbox-performance | 1an | Défini par le plugin GDPR Cookie Consent, ce cookie est utilisé pour stocker le consentement de l'utilisateur pour les cookies de la catégorie " Performance ". |
| CookieLawInfoConsent | 1 an | Enregistre l'état du bouton par défaut de la catégorie correspondante et l'état du CCPA. Il fonctionne uniquement en coordination avec le cookie principal. |
| JSESSIONID | session | Le cookie JSESSIONID est utilisé par New Relic pour stocker un identifiant de session afin que New Relic puisse surveiller le nombre de sessions pour une application. |
| PHPSESSID | session | Ce cookie est natif des applications PHP. Le cookie est utilisé pour stocker et identifier l'ID de session unique d'un utilisateur dans le but de gérer la session de l'utilisateur sur le site Web. Le cookie est un cookie de session et est supprimé lorsque toutes les fenêtres du navigateur sont fermées. |
| viewed_cookie_policy | 1an | Ce cookie est défini par le plugin GDPR Cookie Consent pour stocker si l'utilisateur a consenti ou non à l'utilisation de cookies. Il ne stocke pas de données personnelles. |
| Cookie | Durée | Description |
|---|---|---|
| bcookie | 1 an | LinkedIn définit ce cookie à partir des boutons de partage LinkedIn et des balises publicitaires pour reconnaître l'ID du navigateur. |
| bscookie | 1 an | LinkedIn définit ce cookie pour stocker les actions effectuées sur le site Web. |
| lang | session | LinkedIn définit ce cookie pour mémoriser le paramètre de langue d'un utilisateur. |
| li_gc | 5 mois 27 jours | Linkedin a défini ce cookie pour stocker le consentement du visiteur concernant l'utilisation de cookies à des fins non essentielles. |
| lidc | 1 jour | LinkedIn définit le cookie lidc pour faciliter la sélection du centre de données. |
| pll_language | 1an | Le cookie pll _language est utilisé par Polylang pour se souvenir de la langue sélectionnée par l'utilisateur lorsqu'il revient sur le site Web, et également pour obtenir les informations relatives à la langue lorsqu'elles ne sont pas disponibles d'une autre manière. |
| UserMatchHistory | 1 mois | LinkedIn définit ce cookie pour la synchronisation de l'identifiant LinkedIn Ads. |
| vOTSXIlGdxFUt | 1 jour | Aucune description |
| xwNvEASRYf_ | 1 jour | Aucune description |
| Cookie | Durée | Description |
|---|---|---|
| _ga | 2 ans | Le cookie _ga, installé par Google Analytics, calcule les données relatives aux visiteurs, aux sessions et aux campagnes et assure également le suivi de l'utilisation du site pour le rapport d'analyse du site. Le cookie stocke les informations de manière anonyme et attribue un numéro généré de manière aléatoire pour reconnaître les visiteurs uniques. |
| _ga_ZM94YP9CD3 | 2 ans | Ce cookie est installé par Google Analytics. |
| _gat_gtag_UA_16961320_1 | 1 minute | Défini par Google pour distinguer les utilisateurs. |
| _gid | 1 jour | Installé par Google Analytics, le cookie _gid stocke des informations sur la façon dont les visiteurs utilisent un site Web, tout en créant un rapport analytique des performances du site. Parmi les données collectées figurent le nombre de visiteurs, leur provenance et les pages qu'ils visitent de manière anonyme. |
| AnalyticsSyncHistory | 1 mois | Linkedin a défini ce cookie pour stocker des informations sur l'heure à laquelle une synchronisation a eu lieu avec le cookie lms_analytics. |
| CONSENT | 2 ans | YouTube place ce cookie par le biais des vidéos youtube intégrées et enregistre des données statistiques anonymes. |
| Cookie | Durée | Description |
|---|---|---|
| _fbp | 3 mois | Ce cookie est défini par Facebook pour afficher des publicités sur Facebook ou sur une plate-forme numérique alimentée par la publicité Facebook, après avoir visité le site Web. |
| fr | 3 mois | Facebook définit ce cookie pour afficher des publicités pertinentes aux utilisateurs en suivant le comportement des utilisateurs sur le Web, sur des sites dotés d'un pixel Facebook ou d'un plugin social Facebook. |
| IDE | 1 an 24 jours | Les cookies Google DoubleClick IDE sont utilisés pour stocker des informations sur la façon dont l'utilisateur utilise le site Web pour lui présenter des publicités pertinentes et en fonction du profil de l'utilisateur. |
| test_cookie | 15 minutes | Le test_cookie est défini par doubleclick.net et est utilisé pour déterminer si le navigateur de l'utilisateur prend en charge les cookies. |
| VISITOR_INFO1_LIVE | 5 mois 27 jours | Un cookie défini par YouTube pour mesurer la bande passante qui détermine si l'utilisateur obtient la nouvelle ou l'ancienne interface du lecteur. |
| YSC | session | Le cookie YSC est défini par Youtube et est utilisé pour suivre les vues des vidéos intégrées dans les pages Youtube. |
| yt-remote-connected-devices | jamais | YouTube définit ce cookie pour stocker les préférences vidéo de l'utilisateur qui utilise une vidéo YouTube intégrée. |
| yt-remote-device-id | jamais | YouTube définit ce cookie pour stocker les préférences vidéo de l'utilisateur qui utilise une vidéo YouTube intégrée. |
| yt.innertube::nextId | jamais | Ce cookie, placé par YouTube, enregistre un ID unique pour stocker des données sur les vidéos de YouTube que l'utilisateur a vues. |
| yt.innertube::requests | jamais | Ce cookie, placé par YouTube, enregistre un ID unique pour stocker des données sur les vidéos de YouTube que l'utilisateur a vues. |
| Cookie | Durée | Description |
|---|---|---|
| DEVICE_INFO | 5 mois 27 jours | Pas de description |
| ln_or | 1 jour | Pas de description |
