
CANBIO – Ausbildungsprogramm für Doktoranden
Individuelle Projekte – Krankheitsmonitoring und biologische Netzwerke
Für die systematische Stratifizierung von Patienten und ihres Ansprechens auf Behandlungen werden neue Methoden benötigt, um Escape-Mechanismen von Tumoren, gleich ob intrinsisch oder erworben, zu verstehen und zu überwinden.
Im dritten Arbeitspaket werden neue Ansätze und Methoden zur Untersuchung von Biomarkern für das Monitoring der Escape-Mechanismen von Tumoren erstellt, wobei ein Schwerpunkt auf Flüssigbiopsien liegen wird. Diese verfeinerte Erkennung neuer, spezifischer und empfindlicher Biomarker wird neben der integrativen Datenanalyse auf Systemebene angewendet werden, um das Fortschreiten von Erkrankungen und das Ansprechen auf die Therapie zu modellieren und schließlich vorherzusagen. Dieses Wissen wird die Grundlage für neue Entdeckungen und technische Anwendungen legen.
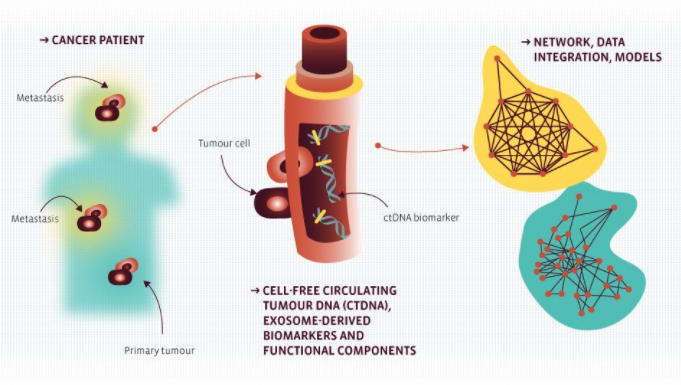
Projekt 1: Zirkulierende tumorabgeleitete DNA und miRNAs: Krebs-Biomarker bei Melanomen und anderen soliden Tumoren
- Betreuung: Dr. Stephanie Kreis (UL)
Zellfreie Tumor-DNA (cfDNA) und auch miRNAs sind interessante Moleküle, die Hinweise auf das Fortschreiten der Erkrankung und/oder die Entwicklung einer Arzneimittelresistenz liefern können. Das Projekt wird beide Molekülklassen in patientenabgeleiteten Proben aus Melanomen analysieren, die während der Behandlung mit Kinaseinhibitoren oder einer Immuntherapie (sofern verfügbar) nachverfolgt werden, um auf diese Weise Biomarker zu identifizieren, mit denen Progression, das Ansprechen auf die Behandlung und die Entwicklung einer Resistenz vorhergesagt werden können, damit Behandlungspläne rechtzeitig geändert werden können. Den Kern des Projekts werden die Isolierung und Profilierung sowohl von cfDNA als auch miRNAs mit anschließender bioinformatischer Analyse und Validierung bilden.
Projekt 2: Exosomproteine und RNA als Biomarker für die Entwicklung und Progression hämatologischer Malignitäten
- Betreuung: Dr. Bassam Janji (LIH/UPS) , Dr. Etienne Moussay (LIH)
Tumorzellen sekretieren kleine Vesikel, sogenannte Exosomen, die an der Kommunikation zwischen Tumor und Stroma sowie an der Entwicklung und Progression einer Erkrankung beteiligt sind. Tumorabgeleitete Exosomen enthalten eine Vielzahl an Proteinen, Nukleinsäuren und Lipiden und sind in allen Körperflüssigkeiten vorhanden. Als vielversprechende Biomarker für die Diagnose und Prognose von Tumoren haben Exosomen viel Aufmerksamkeit erfahren. Ziel dieses Projekts ist es, potenzielle Biomarker in Exosomen zu identifizieren, die aus präklinischen Mausmodellen und Zellen von Patienten abgeleitet wurden, welche von hämatologischen Malignitäten betroffen sind. Darüber hinaus soll der Einfluss dieser Exosomen auf Zellen aus der Mikroumgebung getestet werden. Kandidaten mit dem größten Potenzial für eine Nutzung in der personalisierten Medizin sollen in Patientenkohorten validiert werden.
Projekt 3: Das Proteom von Exosomen als Quelle nichtinvasiver Biomarker bei soliden Tumoren
- Betreuung: Dr. Gunnar Dittmar (LIH), Dr. Guy Berchem (LIH/CHL)
Da sie die Vorteile einer nichtinvasiven Probenentnahme und die Verfügbarkeit von somatischen Veränderungen in sich vereinen, werden Flüssigbiopsien, die vom Patientenblut gereinigte Exosomen verwenden, zunehmend als alternative Lösung für die Diagnostik untersucht. In diesem Projekt wird der Doktorand/die Doktorandin einen auf Flüssigchromatographie und Massenspektrometrie (LC-MS) basierten Proteomik-Ansatz entwickeln, um Proteinbiomarker von soliden Tumoren in Plasmaexosomen im Vergleich zu Gewebebiopsien zu analysieren. Zusätzlich zur Aktivierung somatischer Mutationen von Krebstreibern werden auch posttranslationale Modifikationen als Biomarker erforscht.
Projekt 4: Entwicklung von Methoden für das Monitoring der Krebsprogression und die Vorhersage des Ansprechens auf eine Behandlung
- Betreuung: Prof. Thomas Sauter (LIH), Prof. Simone Niclou (LIH)
Das Projekt wird neue rechnergestützte Modelle für die Vorhersage des Ansprechens auf eine Behandlung entwickeln. Dies soll durch die integrierte Analyse verschiedener Typen von „Omik“-Datensätzen erreicht werden. Der/die erfolgreiche Bewerber/in wird unter den zu implementierenden Modellierungsstrategien Assoziationsnetzwerke biologischer Daten für die Patientenstratifizierung aufbauen und analysieren. Das Projekt wird verschiedene Anwendungen untersuchen, wobei die Forschung zu Glioblastomen im Mittelpunkt stehen wird. Dabei ist eine intensive Zusammenarbeit mit dem Kocher’s Lab an der Mayo Clinic in den USA vorgesehen.
Projekt 5: Nutzung der Fähigkeit zur Generierung von Multi-Omik-Daten und systembasierte Modellierungsansätze für die Untersuchung der Escape-Mechanismen von Tumoren
- Betreuung: Prof. Thomas Sauter (UL)
Die überwältigende Menge an Daten erfordert effiziente rechnergestützte Werkzeuge, welche die Integration dieser Daten und eine erkrankungs- (patienten-)spezifische Netzwerkanalyse ermöglichen. Wir werden unsere Algorithmen für die Rekonstruktion metabolischer Netzwerke weiterentwickeln und einen Arbeitsablauf für die Identifizierung von Wirkstoff-Targets auf der Grundlage karzinomspezifischer Modelle metabolischer Netzwerke erstellen. In Zusammenarbeit mit Prof. Dagmar Kulms von der Arbeitsgruppe experimentelle Dermatologie am Universitätsklinikum Dresden werden wir diese neue Methode an einem Panel von Melanomzelllinien validieren.
Projekt 6: Zirkulierende Tumor-DNA: Beurteilung unterschiedlicher molekularer Strategien für einen extrem empfindlichen Nachweis erworbener somatischer Veränderungen
- Betreuung: Prof. Michel Mittelbronn (LNS), Dr. Daniel Stieber (LNS)
Aus Plasma abgeleitete zellfreie Tumor-DNA (ctDNA) bietet sich als möglicher Ersatz für aus Gewebe abgeleitete Tumor-DNA an. Durch eine Analyse der ctDNA können Tumoren mit einer nichtinvasiven Methode erkannt und überwacht werden, ohne dass Proben wiederholt invasiv entnommen werden müssen. Zudem können hiermit potenziell umfassende Befunde der vollständigen genetischen Heterogenität innerhalb des Tumors erfasst werden. Um dieses Ziel in einer klinischen Diagnoseumgebung zu verwirklichen, muss eine sehr hohe analytische Sensitivität erreicht werden. Ziel des Projekt ist es, unterschiedliche, extrem empfindliche molekulare Strategien auf ihre Fähigkeit hin zu beurteilen, zuverlässig wiederkehrende molekulare Ereignisse in soliden Tumoren in ctDNA nachzuweisen. Der klinische Nutzen dieses Ansatzes wird in einer Pilotstudie validiert, in der das Ansprechen auf die Behandlung und das Fortschreiten der Erkrankung bei Patienten mit nicht-kleinzelligem Lungenkarzinom (NSCLC) überwacht werden.

CANBIO ENTDECKEN
Teilnehmende Forschungsgruppen
- WP1: Intrinsische Fluchtmechanismen
- WP2: Induzierte Fluchtmechanismen
- WP3: Krankheitsüberwachung und biologische Netzwerke
Ausbildung und berufliche Weiterentwicklung
Aufnehmende Institutionen und Partner
Kontakt
Wenn Sie Fragen zu CANBIO haben, wenden Sie sich bitte an:
Prof Simone Niclou
Finanziert durch:

