
CANBIO – Programme de formation doctorale
Projets individuels – Suivi des maladies et réseaux biologiques
Pour comprendre et surmonter les mécanismes d’échappement des tumeurs, qu’ils soient intrinsèques ou acquis, il est indispensable de disposer de nouvelles méthodes de stratification systématique des patients et de leurs réponses thérapeutiques.
Dans le cadre du troisième lot de travaux, de nouvelles approches et méthodologies seront établies pour étudier les biomarqueurs permettant un suivi des mécanismes d’échappement des tumeurs, en mettant l’accent sur les biopsies liquides. Une détection perfectionnée de nouveaux biomarqueurs spécifiques et sensibles ainsi que l’analyse intégrative des données au niveau des systèmes seront appliquées pour modéliser et éventuellement prédire la progression de la maladie et la réponse thérapeutique. Ces connaissances serviront de base à de nouvelles découvertes et applications techniques.
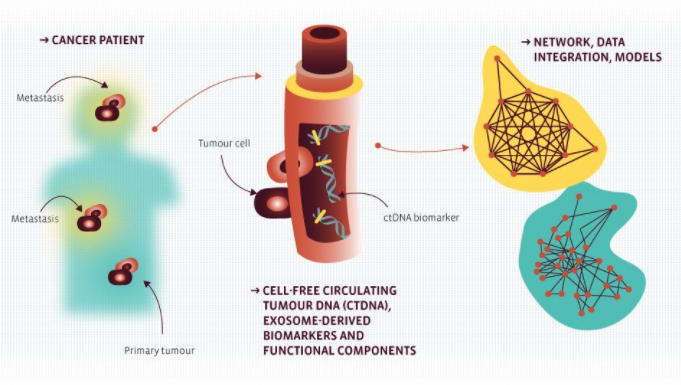
Projet 1 : ADN et micro-ARN circulants dérivés de tumeurs : des biomarqueurs du cancer dans le mélanome et d’autres cancers solides
- Supervision: Dr Stephanie Kreis (UL)
L’ADN acellulaire (cfDNA) et les micro-ARN (miARN) tumoraux sont des molécules intéressantes qui peuvent servir d’indicateurs de la progression de la maladie ou du développement d’une résistance aux médicaments. Le projet analysera ces deux classes de molécules dans des échantillons dérivés de patients atteints de mélanome et suivis lors d’un traitement par inhibiteur de kinase ou par immunothérapie (si disponible) dans le but d’identifier les biomarqueurs qui prédisent la progression, la réponse thérapeutique et le développement de la résistance afin de pouvoir modifier les schémas thérapeutiques à temps. L’isolement et le profilage du cfDNA et des miARN, suivis d’une analyse bioinformatique et d’une validation, seront au cœur du projet.
Projet 2 : protéines et ARN d’exosome comme biomarqueurs du développement et de la progression des hémopathies malignes
- Supervision: Dr Bassam Janji (LIH/UPS) , Dr Etienne Moussay (LIH)
Les cellules tumorales sécrètent de petites vésicules appelées exosomes qui sont impliquées dans la communication avec le stroma tumoral ainsi que dans le développement et la progression de la maladie. Les exosomes dérivés des tumeurs contiennent une variété de protéines, d’acides nucléiques et de lipides et se retrouvent dans tous les fluides corporels. Le rôle prometteur des exosomes en tant que biomarqueurs du diagnostic et du pronostic des tumeurs suscite beaucoup d’intérêt. Ce projet a pour objectif d’identifier des biomarqueurs potentiels dans des exosomes dérivés de modèles précliniques murins et de cellules de patients atteints d’hémopathies malignes et de tester leur influence sur les cellules du microenvironnement. Les biomarqueurs présentant le plus grand potentiel d’utilisation en médecine personnalisée seront validés dans des cohortes de patients.
Projet 3 : le protéome des exosomes comme source de biomarqueurs non invasifs dans les tumeurs solides
- Supervision: Dr Gunnar Dittmar (LIH), Dr Guy Berchem (LIH/CHL)
Combinant les avantages d’une méthode de collecte d’échantillon non invasive et la disponibilité des variations somatiques, les biopsies liquides utilisant des exosomes purifiés issus de sang de patients sont de plus en plus étudiées comme une solution alternative de diagnostic. Dans ce projet, le doctorant développera une approche protéomique basée sur la chromatographie en phase liquide-spectrométrie de masse (LC-MS) pour analyser les biomarqueurs protéiques des tumeurs solides dans les exosomes plasmatiques en comparaison avec les biopsies tissulaires. Outre les mutations somatiques activatrices de pilotes de cancer, les modifications post-traductionnelles seront également explorées en tant que biomarqueurs.
Projet 4 : développement de méthodologies pour le suivi de la progression du cancer et la prédiction de la réponse thérapeutique
- Supervision: Prof Thomas Sauter (LIH), Prof Simone Niclou (LIH)
Ce projet visera à développer de nouveaux modèles informatiques destinés à prédire les réponses thérapeutiques. Et ce par le biais de l’analyse intégrée de différents types d’ensembles de données « omiques ». Parmi les stratégies de modélisation à mettre en œuvre, celle retenue parmi les candidates générera et analysera des réseaux d’association biologique pour la stratification des patients. Les recherches du projet porteront sur diverses applications, avec un accent particulier sur la recherche sur le glioblastome. Il impliquera une forte coopération avec le laboratoire du Dr Kocher à la Mayo Clinic aux États-Unis.
Projet 5 : exploitation de la capacité de production de données multi-omiques et des approches de modélisation basées sur les systèmes pour étudier les mécanismes d’échappement des tumeurs
- Supervision: Prof Thomas Sauter (UL)
Les quantités considérables de données nécessitent des outils informatiques efficaces qui permettent l’intégration et l’analyse de réseaux spécifiques aux maladies (ou aux patients). Sur base de modèles de réseaux métaboliques spécifiques au cancer, nous poursuivrons le développement de nos algorithmes de reconstruction des réseaux métaboliques et établirons un flux de travail en vue de l’identification de cibles médicamenteuses. Nous validerons cette nouvelle méthodologie sur un panel de lignées cellulaires de mélanome en collaboration avec la professeure Dagmar Kulms, du groupe de dermatologie expérimentale de l’University Hospital de Dresde.
Projet 6 : ADN tumoral circulant : évaluation de différentes stratégies moléculaires en vue de parvenir à une détection ultrasensible des altérations somatiques acquises
- Supervision : Prof Michel Mittelbronn (LNS), Dr Daniel Stieber (LNS)
L’ADN tumoral circulant (ctDNA) dérivé du plasma a démontré son potentiel en tant que substitut de l’ADN tumoral dérivé des tissus. L’analyse du ctDNA peut servir à la détection et au suivi non invasifs des tumeurs en éliminant la nécessité de répéter les prélèvements d’échantillons invasifs. Elle possède en outre le potentiel de saisir de manière exhaustive l’hétérogénéité génétique intratumorale. Cependant, pour atteindre cet objectif dans le cadre d’un diagnostic clinique, il faut atteindre une sensibilité analytique très élevée. Le but du projet est d’évaluer de manière comparative différentes stratégies moléculaires ultrasensibles en ce qui concerne leur capacité à détecter de manière fiable les événements moléculaires récurrents dans les tumeurs solides à l’aide du ctDNA. L’utilité clinique de cette approche sera validée dans une étude pilote de suivi de la réponse thérapeutique et de la progression de la maladie chez des patients atteints de cancer bronchique non à petites cellules (CBNCP).

DéCOUVRIR CANBIO
Groupes de recherche participants
- WP1: Mécanismes d’évasion intrinsèques
- WP2: Mécanismes d’évasion induits
- WP3 Surveillance des maladies et réseaux biologiques
Formation et développement de carrière
Institutions et partenaires impliqués
Contact
Pour toute question relative à CANBIO, veuillez contacter :
Prof Simone Niclou
Financé par :

